CINETICA ENZIMATICA
Sappiamo che una reazione chimica è una trasformazione cui va incontro una specie chimica (reagente) che modifica la propria struttura e composizione per creare una o più specie chimiche diverse da quella originaria (prodotto).Le reazioni chimiche non modificano la natura degli atomi singoli o legati tra loro nelle molecole, ma solo il modo con cui sono legati. Per esempio, la specie HI acido iodidrico si scinde per formare H ed I secondo la reazione 2HI→ H2 +I2 in cui la freccia indica il verso in cui procede la reazione. La sostanza reagente viene consumata nel tempo e contemporaneamente si ottengono i prodotti. La velocità con cui i reagenti diminuiscono la loro quantità (espressa sempre come concentrazione) nel tempo, viene definita velocità di reazione. Esistono reazioni velocissime talmente veloci da essere incontrollabili o addirittura esplosive e reazioni lentissime tanto da non poter essere utilizzate ma tutte quante sono regolate da fattori diversi quali Temperatura, Pressione (solo per i gas) ,Concentrazione, Capacità dei reagenti di entrare in contatto tra loro. Secondo la Teoria delle Collisioni, una reazione avviene in seguito agli urti fra le molecole. Durante il corso di una reazione, si rompono dei legami e se ne formano di nuovi e quindi risulta evidente che solo una frazione degli urti possiede energia sufficiente poter reagire. Perché gli urti siano efficaci, gli atomi o molecole devono possedere un’energia superiore ad una limite minimo detto Energia di Attivazione. Se l’energia di attivazione della reazione è alta, si verificano pochi urti e la velocità di reazione è bassa, viceversa se la barriera è bassa la velocità è alta. In realzione alla teoria degli urti è chiaro che un aumento di temperatura provoca l’aumento di velocità degli atomi o molecole aumentando così il numero di urti e quindi la velocità di una reazione. La figura seguente è un grafico che mostra l’avanzamento della reazione (sulle ascisse) e l’energia della molecola(sulle ordinate).

Non tutta la molecola di enzima è interessata alla reazione ma solo uno o più punti di essa chiamati “siti attivi” che non sono altro che delle zone ben definite della struttura che sono attivamente implicate nel processo di catalisi.Le proprietà chimiche e la struttura spaziale del sito attivo di un enzima permettono il riconoscimento del substrato ed il successivo legame specifico. Il sito attivo può essere considerato come una piccola concavità posta sulla superficie dell’enzima all’interno della quale vi sono dei punti caratterizzati da una carica oppure idrofobicità oppure ancora substrutture streriche responsabili della specificità. Spesso questi punti agiscono da accettori o donatori di protoni H+o sono legati ad un cofattore quale NAD Piridossale o Tiamina. I cofattori sono infatti piccole molecole di natura non proteica o uno ione di un metallo che si lega all’enzima vicino al sito attivo e ne rende possibile l’attività. In base alla loro natura chimica, i cofattori sono suddivisi in metalli e coenzimi (intesi come piccole molecole organiche).La maggior parte degli enzimi che richiedono il legame a cofattori, infatti, perde ogni funzionalità in caso di loro assenza. Esistono, inoltre, sostanze che inibiscono l’attività enzimatica legandosi direttamente al sito attivo equindi inibendone l’attività.Un enzima privo del cofattore che ne rende possibile l’attività enzimatica è detto apoenzima. Il legame tra cofattore ed apoenzima dà origine al cosiddetto oloenzima (detto anche oloproteina). In genere i cofattori metallici sono legati con legami deboli o con legami covalenti mentre i coenzimi se legati in maniera debole sono detti Coenzimi ma se sono legati con legami forti si definiscono gruppi Prostetici. I coenzimi sono in grado di associarsi e dissociarsi agevolmente dall’enzima, e si trovano associati all’enzima solo se stanno catalizzando una reazione, altrimenti sono dissociati.
Qui di seguito è mostrata la colinesterasi (AChE) un enzima del sistema nervoso. L’acetilcolinesterasi (AChE) è l’enzima chiave nel sistema nervoso degli animali. Mediante idrolisi rapida del neurotrasmettitore, acetilcolina (ACh), AChE termina la neurotrasmissione alle sinapsi colinergiche. È un enzima molto veloce, specialmente per una serina idrolasi, che funziona ad un ritmo che si avvicina a quello di una reazione controllata dalla diffusione. Gli inibitori di AChE sono tra i farmaci chiave approvati dalla FDA per la gestione della malattia di Alzheimer (AD). La potente tossicità dei veleni organofosforati (OP) o dei gas nervini è attribuita principalmente ai loro potenti inibitori dell’AChE.

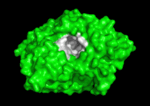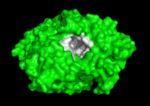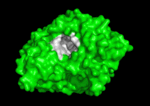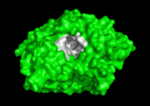
Meccanismo di azione:
I substrati si legano al sito attivo dell’enzima con legami idrogeno, interazioni idrofobiche, legami covalenti temporanei o una combinazione di questi. I residui del sito attivo agiscono da donatori o accettori di protoni o altri gruppi presenti sul substrato favorendo la reazione. In questo modo il sito attivo modifica il meccanismo di reazione secondo un percorso al quale è associata una minore energia di attivazione. Il prodotto legato al sito attivo è solitamente instabile a causa dell’impedimento sterico e quindi tende a essere liberato ricostituendo in tal modo l’enzima originario. Due sono i modelli proposti su come il sito attivo dell’enzima si adatti al suo specifico substrato:
- modello della chiave-serratura
- ipotesi dell’adattamento indotto.
meccanismo chiave serratura

ipotesi dell’adattamento indotto.
Il modello chiave serratura in realtà è un modello in cui i siti attivi sono abbastanza rigidi,tuttavia, dal momento che in realtà gli enzimi sono strutture relativamente flessibili nel 1958 Daniel Koshland ha proposto una modifica del modello chiave-serratura suggerendo che il sito attivo potesse continuamente modellarsi in base alla presenza o meno del substrato.Il substrato pertanto non si lega ad un sito attivo rigido, ma provoca un rimodellamento sterico del sito stesso, che determina un legame più stabile dell’enzima in modo da portare correttamente a termine la sua attività catalitica. In alcuni casi, ad esempio con la glicosidasi, anche il substrato può cambiare leggermente la propria forma all’ingresso nel sito attivo.
Specificità degli enzimi
La maggior parte degli enzimi presenta una notevolissima specificità per la reazione catalizzata e per i substrati coinvolti. Tale specificità è legata a diversi fattori che caratterizzano l’associazione tra il substrato e il sito attivo, come la complementarità dal punto di vista strutturale, le cariche elettriche, la natura idrofoba o idrofila. Gli enzimi mostrano spesso livelli elevatissimi di stereospecificità, regioselettività e chemioselettività.
Alcuni degli enzimi che mostrano la maggiore specificità sono coinvolti nella replica ed espressione del genoma. Enzimi come le DNA polimerasi catalizzano inizialmente la reazione di allungamento del DNA, quindi valutano in un secondo momento l’efficienza e la correttezza dell’operazione stessa. Questo processo in due passaggi permette di ridurre enormemente gli errori compiuti Simili meccanismi detti proof-reading sono presenti anche nelle RNA polimerasi, aminoacil-tRNAsintetasi e nei ribosomi. Esistono, in ogni caso, diversi enzimi caratterizzati da una specificità relativamente più bassa e che sono in grado di agire su un numero ampio di substrati.
CINETICA ENZIMATICA
Vista la notevole importanza degli enzimi nei processi biochimici degli esseri viventi occorre studiare la velocità con cui avvengono le reazioni enzimatiche in quanto ci permettono di valutare la quantità dei substrati analizzati. Consideriamo la generica reazione
A→ B
Questa equazione ci dice che A dà origine al prodotto B. Poiché siamo in grado di misurare con metodiche analitiche le quantità dei partecipanti alla reazione (A, B),vediamo che la concentrazione di A diminuisce col passar del tempo, mentre quella di B aumenta.
La variazione di concentrazione di un reagente o di un prodotto in un certo intervallo di tempo non è altro che la velocità con cui il reagente si consuma ed i prodotti si formano in quell’intervallo di tempo cioè la VELOCITA’ DI REAZIONE.
Matematicamente la diminuzione della concentrazione del reagente A è espressa da:
V= -d[A]/t oppure V= d[B]/t il segno meno indica che [A] diminuisce nel tempo t.
Poichè la velocità dipende dalla prima potenza della concentrazione di A cioè [A]1 la reazione viene definita del PRIMO ORDINE. Nel caso in cui si ha ad esempio la reazione
2A → B
la velocità sarà V=k [A]2 e la reazione è definita del secondo ordine. Più in generale se abbiamo una reazione
mA + n B → x C
la reazione dipende da [A]m[B]n ed in tal caso l’ordine della reazione è dato da m+n.
Cinetica enzimatica ad un solo substrato.
La prima equazione generale di velocità per una reazione enzimatica fu derivata nel 1903 da Victor Henri. L’equazione di Henri era in grado di spiegare l’ osservazione che la velocità iniziale della reazione enzimatica è direttamente proporzionale alla concentrazione dell’enzima tuttavia essa cresce in modo non lineare al crescere della concentrazione del substrato fino ad un valore massimo limite. Gli studi vennero ripresi da Leonor Michaelis e Maud Menten nel 1913 da cui ha preso il nome l’equazione relativa alla reazione ad un substrato.Il principio di base del ragionamento di Michaelis e Menten è che il substrato S e l’enzima E formino un complesso ES in una reazione in equilibrio e che una volta formatosi il complesso si scinde per formare il prodotto ripristinando l’enzima:
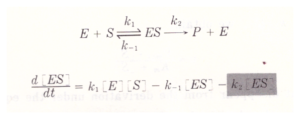
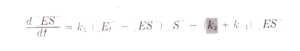
essendo la concentrazione totale di E [Et]= [E]+[ES] allora [E]= [Et] – [ES]
quindi
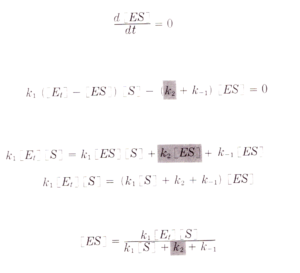
e dividendo numeratore e denominatore per K1 si ha:
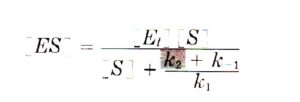
sapendo inoltre che V= k2[ES] si ha
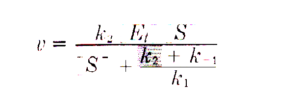
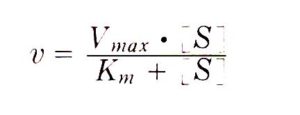
dove Km= k2+k-1/k1 e Vmax= K2 [Et]
Il grafico seguente mostra come varia la concentrazione di E,ES, S e P col passare del tempo:
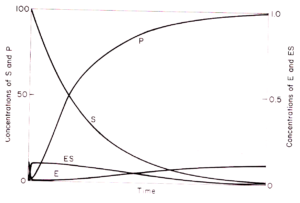
La velocità di variazione della [E] e di [ES] col tempo è minima se paragonata alla velocità di variazione di [S] e di [P] confermando il lavoro di Briggs ed Haldane noto come Steady State Approssimation: immediatamente dopo l’inizio della reazione enzimatica, viene raggiunto uno stato stazionario in cui la velocità di variazione della concentrazione dell’ intermedio ES è essenzialmente zero se paragonata alla velocità di variazione della concentrazione dei reagenti e dei prodotti. In termini matematici
l’ espressione
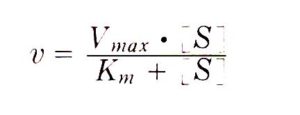
può essere semplificata se [S] è molto più grande di Km così che il contributo di Km può essere trascurato per cui
V= K [Et] cioè la velocità della reazione enzimatica è direttamente proporzionale alla quantità di enzima totale solo se [S] è molto grande.
L’equazione di Michaelis può essere riscritta V= Vmax/ Km/[S] +1
Sia Km che Vmax sono caratteristiche per ogni reazione enzimatica e conoscendo questi valori possiamo calcolare la velocità della reazione per un intero range.
Se poniamo [S] = Km allora V= Vmax [S] /[S]+[S] V= Vmax /2 per cui possiamo dire che:
Km è“la concentrazione di substrato che dimezza la velocità della reazione enzimatica”.
Riassumendo:
V= velocità della reazione enzimatica
[S] = concentrazione di substrato
Km= la [S] che riduce della metà la velocità massima della reazione
Vmax velocità massima che può raggiungere la reazione anche se variamo la [S]
Vmax si esprime in moli/minuto di [S] per mole di enzima cioè mole/mole x minuto= 1/minuto oppure minuto-1
Inoltre ricordando che Vmax =K2 [Et] se [Et] = unità molare allora Vmax= K2
Vmax in questo caso rappresenta il numero di molecole catalizzate dall’unità di enzima al minuto.
Se usiamo basse concentrazioni di substrato tali che [S] << Km cioè se [S] è almeno Km/10 l’espressione V= Vmax[S] /km+ [S]
allora V = Vmax [S]/Km e siccome Vmax/Km = costante ciò significa che
la velocità è direttamente proporzionale alla [S] quando [S] <<Km
Ciò vuol dire che possiamo valutare la quantità di substrato da misure di velocità.
Se al contrario [S]>>Km oppure se [S]= almeno a 10 Km allora Km è trascurabile V= Vmax [S]/[S]
Quindi V= Vmax il che significa che è possibile quantificare l’enzima da misure di velocità.
COME DETERMINARE I VALORI DI Km E Vmax GRAFICAMENTE
a questo scopo utilizziamo un esempio pratico:
Supponiamo che il GLUCOSIO si trasformi in un altra sostanza mediante un enzima e misuriamo la velocità di reazione nei seguenti casi
| concentrazione glucosio M mmoli/l convertiti al minuto
per ogni mole di enzima /litro ______________________________________________________ 1 x 10 -5 150 2x 10-5 256 1 x 10-4 600 3 x 10-4 800 5 x 10-4 818
|
da questi dati costruiamo un grafico velocità concentrazione
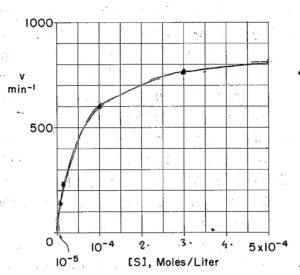
da questa curva, che è un’iperbole rettangolare, si vede che la velocità massima si può valutare solo approssimativamente per cui è più agevole il calcolo se questa iperbole viene trasformata in linea retta la cui espressione matematica è del tipo Y=a+ bX .
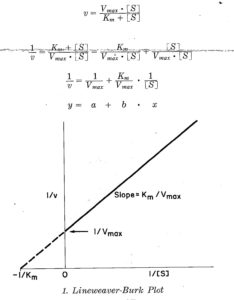
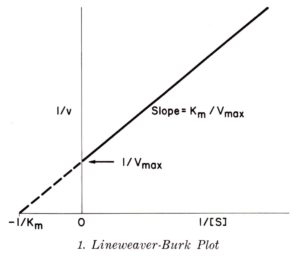
LINEARIZZAZIONE SECONDO LINEWEAVER E BURK
Lineweaver e Burk hanno effettuato la trasformazione nel seguente modo:
LINEARIZZAZIONE DI WOOLF
Il metodo di woolf è il seguente:
dall’equazione di Lineweaver e Burk 1/v=1/Vmax +Km/Vmax x 1/[S]
moltiplicando per [S] si ha [S]/V = [S]/Vmax + Km/Vmax
che si può riscrivere [S]/V = Km/Vmax + 1/Vmax [S]
che è della forma Y = a + b X
il grafico che ne risulta è :
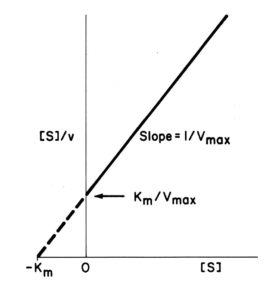
vediamo che il metodo Woolf è molto simile al metodo Lineweaver Burk

un altro metodo è quello di Augustinsson- Hofstee
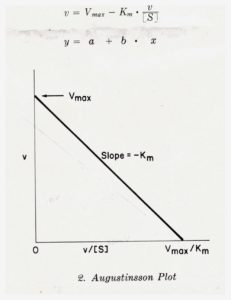
Utilizzando il metodo Lineweaver e Burk per il nostro esempio del Glucosio avremo

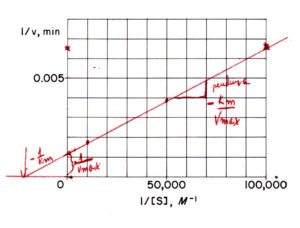
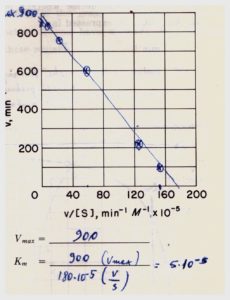
Si può notare che a concentrazioni dell’ordine di 10-5 con questo metodo si ha una certa aberrazione per cui molti preferiscono usare il metodo Augustinsson o Woolf che nel caso specifico mostra il risultato seguente:

UN ESEMPIO DI NON CORRISPONDENZA ALL’EQUAZIONE DI MICHAEILS MENTEN
Osserviamo la curva sperimentale seguente:
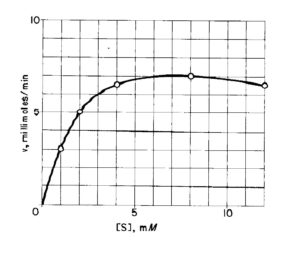
Potrebbe a prima vista apparire che la cinetica sperimentale corrisponda all’equazione di Michaelis Menten, tuttavia se linearizziamo la curva secondo Lineweaver Burk otteniamo
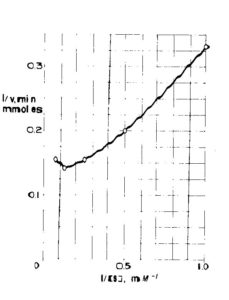
questo denota che la reazione enzimatica è più complessa che non quella del glucosio che abbiamo preso come esempio e cioè
E+S <=> ES → E + P
Per poter spiegare la forma di questa curva, è plausibile pensare che probabilmente un eccesso di substrato abbia inibito la reazione in qualche modo.Osserviamo adesso la reazione tra enzima e due diversi zuccheri :glucosio e mannosio e consideriamo i dati che ne risultano:
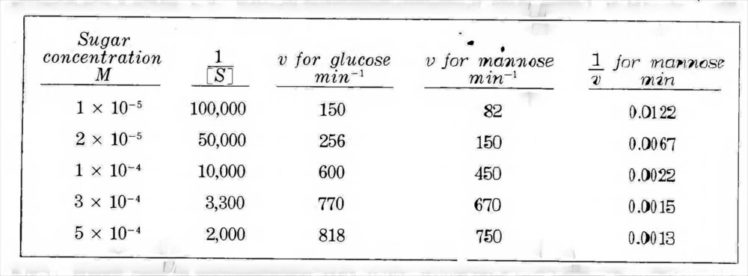
mettendo in grafico la velocità in funzione della concentrazione di glucosio e quella del mannosio otteniamo le due curve
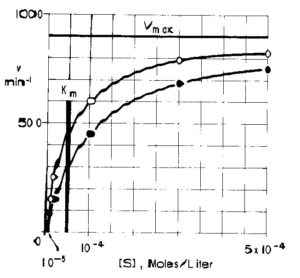
questo grafico suggerisce che la Vmax del mannosio è simile a quella del glucosio. Se mettiamo in grafico il reciproco della concentrazione di mannosio otteniamo
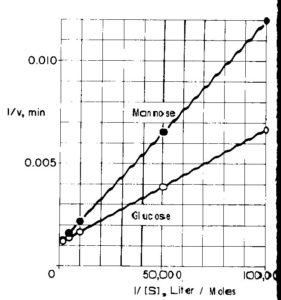
da cui possiamo calcolare i valori di Vmax e Km per il mannosio:
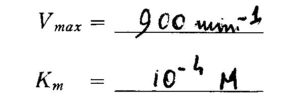
Si ottiene perciò il Numero di Turnover che è il valore di Vmax quando la concentrazione dell’enzima totale è uguale ad 1 mole in base a ciò
Vmax= k2 [E] da cui Vmax =K2 = 900 min-1
se consideriamo adesso il grafico
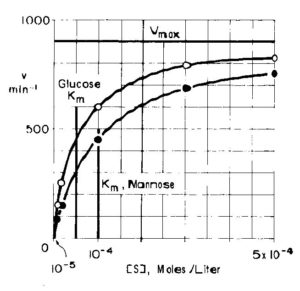
Può sembrare strano che il substrato che reagisce più lentamente abbia un valore di Km più alto, ma si vede che per produrre la stessa velocità semi-massima, si richiede un livello di mannosio due volte più alto, quindi, il mannosio deve essere il substrato meno reattivo.
Occorre sempre tener presente che, mantenendo invariati gli altri fattori della reazione enzimatica, più alto è il valore di Km più grande deve essere la concentrazione di substrarto per ottenere la velocità semimassima e quindi minore è la reattività del substrato.
Per esempio se consideriamo l’ornitina ad una concentrazione C=10-4 M e se supponiamo che reagisca con l’enzima A ed abbia Km= 10-3 M e con un secondo enzima B per cui Km=10-5 M e se supponiamo valori di Vmax simili per i due enzimi allora l’attacco avviene principalmente da parte dell’enzima B perchè più bassa è la concentrazione di substrato maggiore è la reattività mentre un aumento di ornitina (quindi della sua concentrazione) produce un valore di Km maggiore e quindi una minore reattività.
COMPORTAMENTO DI DUE SUBSTRATI CHE COMPETONO PER LO STESSO ENZIMA
Supponiamo che mannosio e glucosio reagiscano con lo stesso enzima e che, al contrario di quanto considerato prima, si trovino ambedue nella stessa soluzione e che reagiscano con un solo enzima E. Ovviamente, anche se V=k2 [Etot] non ci possiamo aspettare che ambedue i substrati reagiscano come se essi fossero da soli nella soluzione. Infatti abbiamo due complessi enzima-substrato:
E + S <=> ES → E+P1
E + S <=> ES → E+P2
e la presenza di uno zucchero influenza la reazione dell’alro con l’enzima.Diciamo allora che il mannosio compete con il glucosio per il sito dell’enzima e viceversa. Si dice che in tal caso si ha una INIBIZIONE COMPETITIVA.
L’inibizione competitiva, può essere superata se si aumenta notevolmente uno dei substrati.Per esempio, nel caso si debba studiare il glucosio in presenza di mannosio, basta aumentare in modo notevole la quantità di glucosio così che tutto l’enzima sarà sotto forma di complesso enzima -glucosio e viceversa se occorre studiare il mannosio. In definitiva un inibitore enzimatico è un composto in grado di legarsi all’enzima diminuendone così l’ attività ma senza essere in grado di fornire prodotti e la loro azione può essere persistente :
E +I <=> EI
Un esempio tipico è la deidrogenasi che catalizza la reazione succinato→ fumarato in presenza di malonato che può esso stesso reagire deidrogenandosi. Il malonato è quindi un inibitore della reazione succinato→fumarato che però non si deidrogena formando il prodotto, blocca solo la reazione.
Per descrivere l’attività inibitoria è stata introdotta una costante Ki analoga a Km e proprio dalle misure di Ki si possono ottenere dati sulla relazione struttura-attività inibitoria. Infatti si considera una molecola inibitoria e si valuta la corrispondente Ki quindi si modifica la struttura molecolare e si ricalcola Ki ed in tal modo si riesce a comprendere la natura sterica del sito attivo dell’enzima. Quando l’enzima si lega all’inibitore può trasformare alcuni residui amminoacidici che si trovano nel sito attivo dell’enzima che sono vitali per l’attività enzimatica.Questo processo è irreversibile mentre esistono inibitori che formano invece un legame non covalente e producono diversi tipi di inibizione a seconda che si leghino all’enzima, al complesso enzima-substrato, o ad entrambi e vengono definiti reversibili. Agli enzimi possono legarsi anche attivatori enzimatici cioè molecole che non sono indispensabili per la catalisi, ma la loro presenza la facilita la reazione enzimatica. Esistono anche i cosiddetti cofattori cioè ioni metallici come calcio, ferro, manganese e coenzimi come prodotti che derivano da vitamine, che invece sono indispensabili per la reazione e la loro assenza non rende possibile il meccanismo enzimatico. Alcuni inibitori enzimatici possono bloccare cofattori e coenzimi per ridurre l’attività dell’enzima.
COME VALUTARE SE UN INIBITORE E’ COMPETITIVO O NON COMPETITIVO
Si misura la velocità di reazione V utilizzando diverse concentrazioni di Substrato e mantenendo costante la concentrazione dell’inibitore.
nell’inibizione competitiva:
Poichè la [ES] tende a rimanere costante durante il periodo di osservazione, è ancora valida la Lineweaver e Burk che sarà sempre una linea retta. Poichè l’effetto competitivo è annullato quando la concentrazione di S è infinita, ne consegue che il valore di Vmax sarà la stessa di quella misurata in assenza di inibitore , questo significa che l’intercetta sull’asse Y in presenza di eccesso di substrato ed in presenza di inibitore sarà identica a quella relativa al solo substrato senza inibitore. Poiché inoltre siamo in presenza di alte quantità di substrato il valore di Km aumenterà rispetto al valore ottenuto senza inibizione e con quantità di substrato non eccessive.
quindi per inibitori competitivi : Vmax rimane costante e Km aumenta
Nel caso degli inibitori non competitivi si può immaginare che essi si leghino all’enzima in modo permanente facendo così diminuire la quantità di enzima presente e questo porta ad un valore di Vmax inferiore rispetto a Vmax della reazione enzima substrato senza inibitore.Inoltre, poichè la quantità di enzima che rimane reagisce normalmente col substrato e poichè Km non dipende dalla concentrazione dell’enzima ne consegue che Km rimane invariato.
Nelle reazioni non-competitive : Vmax diminuisce Km rimane costante
Riassumendo pertanto si ha:
tipo di inibizione effetto su Vm effetto su Km
competitiva Vm invariata Km aumentata
non competitiva Vm diminuita Km invariata
________________________________________________
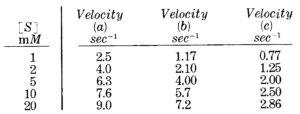
La tavola seguente mostra le velocità calcolate per una reazione enzimatica con un substrato
a) in assenza di inibitore
b) in presenza di una quantità costante di un inibitore A
c) in presenza di una quantità costante di un inibitore B
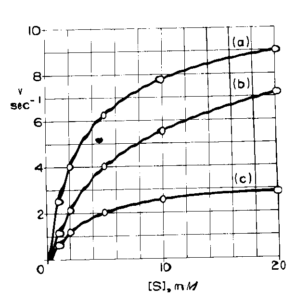
e plottiamo i dati ottenuti
osservando la tavola è probabile che la b) sia una inibizione competitiva e la C) competitiva, ma in ogni caso non siamo in grado dal grafico ottenuto di definirlo con certezza. Ne consegue che è meglio utilizzare la linearizzazione di Lineweaver e Burk
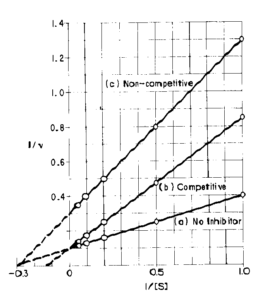
il cui grafico è:
questo grafico è molto più esplicativo in quanto si oserva che in a) e b) Vmax rimane uguale (1/V) mentre Km aumenta (pendenza maggiore) quindi b) rappresenta una inibizione competitiva. C) invece ha un valore di Vmax diminuita e Km costante.
Per quanto riguarda i meccanismi di azione degli inibitori la figura seguente tratta da Wikipedia è molto esplicativa :

Gli inibitori irreversibili modificano l’enzima formando legami covalenti perchè contengono gruppi reattivi come il gruppo CO oppure doppi legami o alogeni che reagiscono con le catene laterali degli amminoacidi. I residui dei siti attivi che vengono modificati sono quelli che presentano catene laterali contenenti gruppi ossidrilici o solfidrici. E’ importante mettere in evidenza che inibire non significa inattivare perché non distruggono la struttura proteica dell’enzima ma modificano solo il sito attivo.L’inattivazione o meglio la denaturazione della proteina che costituisce l’enzima si ha infatti in seguito a trattamenti di modifica dei livelli di PH e temperatura oppure per aggiunta nel mezzo di HCl concentrato caldo, che rompe i legami peptidici della proteina e libera in soluzione gli amminoacidi di cui essa è composta.
Gli inibitori reversibili generalmente non reagiscono con i residui dei siti attivi e quindi possono essere facilmente rimossi per diluizione o mediante dialisi. Abbiamo già discusso degli inibitori enzimatici reversibili che sono
- Inibitori competitivi‘ che competono con il substrato nel legarsi al sito attivo dell’enzima . Essi causano la diminuzione della concentrazione di enzima libero disponibile ma non variano la velocità massima Vmax.
- Inibitori acompetitivi, spesso si legano al complesso enzima-substrato ES. Il legame dell’inibitore influenza il legame del substrato, e viceversa. Questo tipo di inibizione può essere ridotta, ma non bloccata, aumentando la concentrazione del substrato. Questo tipo di inibizione è dovuta al cosiddetto effetto allosterico di cui discuteremo in seguito ed in cui l’inibitore si lega ad un sito differente dell’enzima e produce una modificazione conformazionale dell’enzima che riduce così l’affinità del substrato per il sito attivo e quindi modifica l’ attività dell’enzima.
- Inibitori non competitivi sono molecole capaci di legare sia l’enzima libero che il complesso enzima-substrato. A seconda delle costanti di inibizione Ki e Ki’, che identificano i due equilibri dell’inibitore “I” con l’enzima libero Ki e il complesso Ki’, l’effetto inibitore ha una forza differente. Se l’inibitore ha affinità identica sia per il complesso che per l’enzima l’effetto è quello di modificare sia la velocità di reazione che la quantità di substrato necessaria a saturare l’enzima (da reazione di primo ordine a reazione di ordine zero). Se l’affinità è maggiore nei confronti del complesso enzima substrato rispetto all’enzima libero l’effetto si osserva sul rilascio del prodotto diminuendo l’efficienza dell’enzima. Se invece l’affinità dell’inibitore è maggiore per l’enzima libero, l’effetto è la riduzione dell’enzima libero come si osserva per l’inibizione competitiva
QUANTIFICAZIONE DELL’INIBIZIONE E SIGNIFICATO DI Ki
In caso di inibizione competitiva, è possibile ottenere un’ equazione simile a quella di Michaelis-Menten in cui appare la concentrazione di substrato:
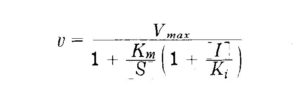
la costante corrispondente che misura la concentrazione di inibitore richiesta per diminuire della metà la velocità di reazione che si ha in assenza di inibitore, viene indicata con Ki.
Se l’inibitore si converte poi in prodotto si ha
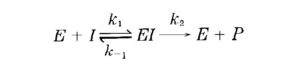
In realtà il valore di Ki non è dato da K2 ma invece rappresenta la costante della reazione di dissociazione di EI K-1 infatti
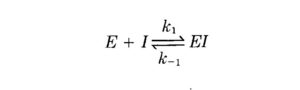
Ricordando che la linearizzazione di Lineweaver e Burk è:
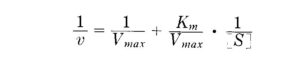
la corrispondente forma della linearizzazione che tiene conto dell’inibitore è :
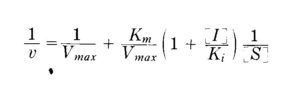
La pendenza in questo c aso è Km/Vmax x [I]/Ki
e confrontandola con la pendenza Km/Vmax x 1/[S] della reazione senza inibitore possiamo calcolare Ki lavorando con una concentrazione [I] nota.
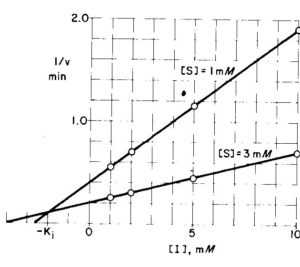
IL GRAFICO DI DIXON PER IL CALCOLO DI Ki
Dixon ha messo in grafico 1/V in funzione di [ I] . Per mostrare il tipo di grafico consideriamo i dati seguenti:

il punto di intersezione delle rette è pari a -Ki in questo caso quindi Ki=òv 2 mM
La costante Ki può essere misurata direttamente con il metodo visto sopra, ma un metodo estremamente accurato è la calorimetria isotermica di titolazione (ITC) che permette di determinare quantitativamente in maniera diretta l’ entalpia di unione di una molecola o di un complesso molecolare.In questo modo è possibile calcolare ΔG della formazione del legame,e di conseguenza ΔS del processo:
- ΔG = –RTlnK = ΔH–TΔS
dove K è la misura diretta dell’affinità di legame Ki, R è la costante dei gas T la temperatura Kelvin ΔH l’entalpia e ΔS l’entropia
REGOLAZIONE CINETICA E BIOLOGICA: FENOMENO ALLOSTERICO
Diversi antibiotici ed alcuni farmaci producono i loro effetti biologici agendo come inibitori di quegli enzimi che sono utilizzati da batteri o virus per infettare le cellule e modificarne il metabolismo. Un razionale utilizzo di certi farmaci prevede l’identificazione dell’enzima implicato in un determinato processo e quindi la sintesi del farmaco che possieda una buona reattività con l’enzima.In questa fase è quindi importante conoscere il valore di Vmax e Ki per il farmaco testato.
E’ ben noto che gli organismi viventi son abili a controllare vari punti del loro metabolismo controllando i metaboliti che si formano utilizzando inibitori o modificatori dell’azione enzimatica. Per esempio, un metabolita può controllare la quantità di produzione di se stesso controllando egli stesso gli enzimi implicati nella sua formazione:questo procedimento è chiamato Inibizione Feedback.
↓‾E1‾E2‾E3↑
A→ B→C→D
Quando la quantità di D supera un certo valore, esso stesso agisce come inibitore dell’enzima E1 facendo quindi diminuire la produzione di D stesso. Consideriamo adesso i seguenti risultati relativi ad una attività enzimatica :
[Substrato] % Velocità Massima
__________________________________
5 6
10 16
20 38
30 58
40 76
50 90
100 97
______________________________
costruiamo adesso il grafico ed otteniamo
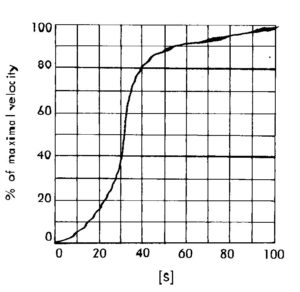
a basse concentrazioni di substrato la velocità aumenta relativamente poco ma a livelli vicini a Km la velocità aumenta molto più rapidamente rispetto a quanto indicato dall’equazione di Michaelis Menten.In questo caso infatti il substrato gioca un doppio ruolo in quanto reagisce con l’enzima nel modo usale, ma si vede anche che al legarsi dalla prima molecola di substrato all’enzima si determina una variazione della sua attività.Questo può causare una diminuzione del valore di Km per il legame della successiva molecola di substrato e come risultato si ha un’accelerazione dell’attività enzimatica.Quando l’enzima mostra questo comportamento allora possiede più di un sito attivo in cui quando il substrato si unisce al primo sito attivo modifica la forma del secondo sito e ne modifica il comportamento. Questo tipo di attività dell’enzima è definito FENOMENO ALLOSTERICO. Essendo il substrato stesso l’agente modificante il fenomeno è detto ALLOSTERISMO OMOTROPICO. In genere allora in questi casi l’enzima deve avere due o più sub unità identiche con attività catalitica ma quando una di esse si lega al substrato modifica la distribuzione sterica dell’altro sito con modifica dell’attività di quest’ultimo.
Pertanto, a basse concentrazioni di substrato, l’azione si svolge essenzialmente attraverso la mediazione di un singolo sito attivo della molecola di enzima. In queste condizioni, dal momento che il valore di Km di questo sito è più grande rispetto a quello dell’ altro sito attivo occupato, la velocità di reazione a questi livelli di substrato è relativamente bassa.
Quando la concentrazione del substrato è abbastanza elevata in modo che la reazione enzimatica si verifichi essenzialmente attraverso l’intermediazione del sito attivo che ha il valore più basso di Km, allora la velocità di reazione sarà relativamente alta.Le proprietà allosteriche di un tale enzima permettono, pertanto, di rispondere alla presenza di una elevata concentrazione di substrato con una maggiore attività. Questo comportamento tende a diminuire considerevolmente il range della variazione di concentrazione del substrato.
La relazione tra la velocità della reazione e la concentrazione del substrato assume la forma di una curva sigmoidale, come abbiamo visto in precedenza quando il substrato ha un effetto allosterico sull’enzima però occorre precisare che la forma sigmoidale della curva può anche avere altre origini.
VISUALIZZAZIONE SPERIMENTALE DELLA FORMAZIONE DI ES
Johnson e Phillips hanno utilizzato la diffrazione ai raggi X ad alta risoluzione dell’enzima Lisozima sotto forma di cristallo, ed hanno fatto un confronto con la stessa metodica dello stesso lisozima dopo esposizione in una soluzione di inibitore competitivo (N-acetilglucosamina) ed hanno osservato che in quest’ultimo caso il pattern mostrava una protuberanza in un punto del lisozima che non era invece presente nella molecola di lisozima iniziale.Questa protuberanza non era altro che la molecola di inibitore legata al lisozima, in quanto altri composti non inibitori non presentavano quel rigonfiamento. E’ stato così individuato il sito attivo della molecola di lisozima.

Lascia un commento